近日,南京医科大学曹晨/杨晟等合作开发并发布了GWAShug,一款旨在系统解读复杂性状的共享遗传基础综合性平台。该平台通过整合大规模的全基因组关联研究(GWAS)数据和计算方法,为复杂性状的共享遗传机制研究提供了标准化工具。相关研究成果发表于国际期刊《Nucleic Acids Research》。
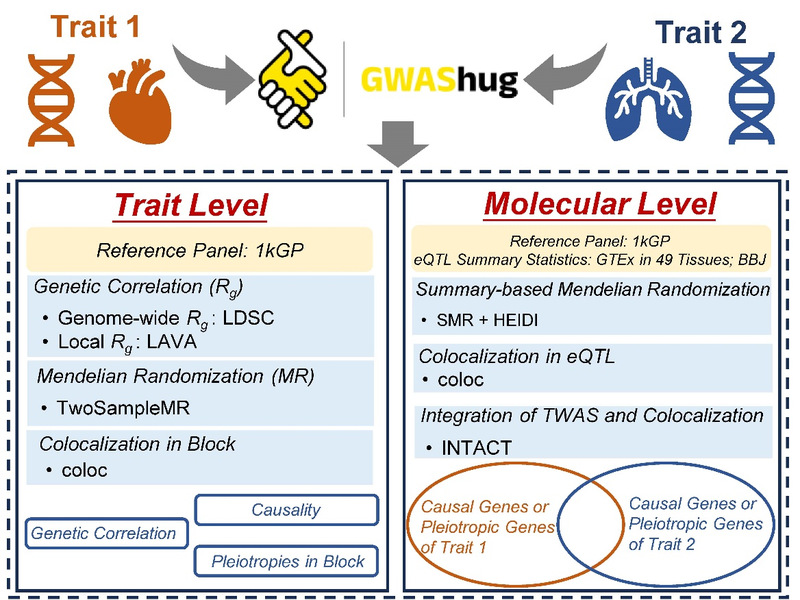
GWAShug平台涵盖了539份高质量GWAS Summary Statistics数据,覆盖296种测量性状和243种疾病性状,囊括了欧洲(EUR)和东亚(EAS)人群的数据资源。平台通过多层次的分析方法,包括遗传相关性、因果关系分析、局部遗传相关性及基因共定位等,为研究者提供从性状到分子水平的全面解读工具。

此外,GWAShug线分析模块支持研究者上传自己的GWAS Summary Statistics数据进行在线分析。该模块配备交互式可视化功能,例如曼哈顿图和LocusZoom图,帮助研究者动态探索复杂性状间的共享遗传基础并生成可下载的分析报告。这种灵活性使得GWAShug不仅适用于生物医学领域的基础研究,还能支持临床遗传学应用。
GWAShug已成功应用于肥胖与心血管疾病共享遗传基础的研究中,发现了多个关键基因(如KCNK3)及潜在的致病机制,为理解多病共存的遗传病因和精准医学策略的开发提供了新方向。其标准化的分析流程和丰富的数据资源,使其成为当前遗传研究领域的重要工具。
生物医学工程系曹晨教授、公共卫生学院黄鹏副教授、杨晟副教授是本研究的共同通讯作者,生物医学工程系硕士研究生田敏、李正晖为研究共同第一作者。研究访问地址:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkae873/7815980?login=false
