
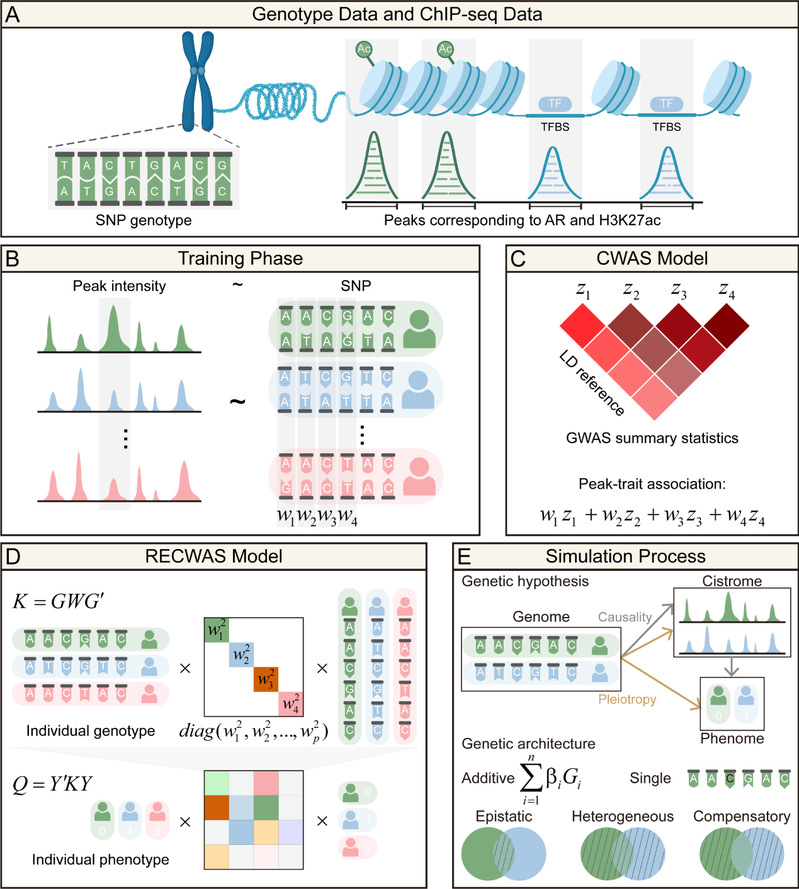
顺反组(cistrome)是全基因组范围内反式作用因子的顺式作用靶点集合,包括转录因子结合位点和组蛋白修饰的位置。全顺反组关联分析(Cistrome-Wide Association Study, CWAS)是一种利用参考基因组和顺反组数据集训练预测模型的方法,通过独立数据集预测顺反组水平,并将其与表型组关联,最终识别与复杂疾病相关的顺反子区域。CWAS已成功应用于前列腺癌等疾病的研究中,识别出疾病易感顺反子风险区域。
近日,南京医科大学生物医学工程与信息学院电磁生物环境与医学先进诊疗技术实验室曹晨教授、顾宁教授提出了一种基于核方法的全顺反组关联分析算法(RECWAS)。该算法通过核方法捕获单核苷酸多态性(SNP)之间的非线性效应,显著提升了CWAS关联算法的统计效能。在真实和模拟的前列腺癌数据中应用后,RECWAS能够识别出更多且更准确的疾病易感风险顺反子区域。这些区域通常是CWAS方法未能识别的,但却与前列腺癌等疾病发病机制有显著关联。多项实验验证了RECWAS在捕获关联风险顺反子区域的准确性及其在前列腺癌和其他疾病中的生物意义。
这项研究的共同第一作者为实验室博士生邵梦婷、硕士生田敏和陈开阳,以及浙江大学的蒋杭进研究员,曹晨教授和顾宁教授为该研究共同通讯作者。
该研究成果已被《Advanced Science》期刊接收并在线发表。文章题为《Leveraging Random Effects in Cistrome-Wide Association Studies for Decoding the Genetic Determinants of Prostate Cancer》,文章访问地址为:https://doi.org/10.1002/advs.202400815。
